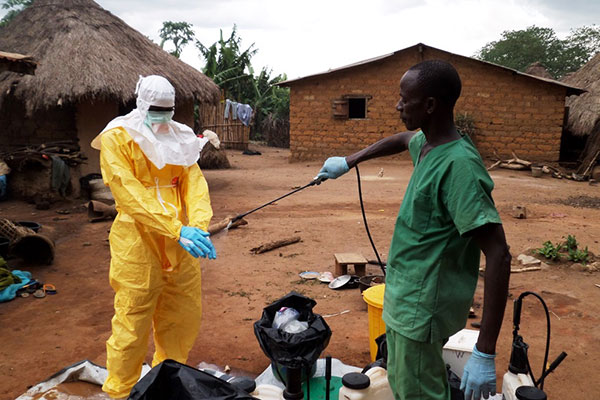
Como resposta ao maior surto de ebola da história, um grupo internacional de cientistas sequenciou e analisou 99 genomas do vírus. Com o estudo, publicado nesta sexta-feira, 29, na revista Science, foi possível rastrear a origem e transmissão do vírus no surto atual – informações que são essenciais para o desenvolvimento de vacinas, diagnósticos e tratamentos. A pesquisa mostrou que o genoma do vírus atual tem mais de 300 modificações genéticas em relação às linhagens das epidemias anteriores da doença.
O estudo foi realizado pelo Broad Institute do MIT e Harvard, em colaboração com o Ministério da Saúde de Serra Leoa. Cinco dos 58 autores do artigo da Science contraíram o vírus do ebola e morreram antes da publicação, entre eles o médico Sheik Humarr Khan, especialista na febre de Lassa – uma febre hemorrágica viral – do Centro Africano de Genômica de Doenças Infecciosas, Saúde Humana e Hereditariedade.
Primeiro surto
Segundo os autores, as linhagens de ebola atuais têm um ancestral comum com o primeiro surto da história, ocorrido em 1976. Os pesquisadores traçaram o caminho de transmissão e as relações evolutivas das amostras, revelando que a linhagem atual divergiu da versão do vírus nos últimos dez anos. Segundo eles, o surto de 2014 teve origem na Guiné e se espalhou por Serra Leoa, Libéria e Nigéria.
De acordo com o artigo, nas ocorrências anteriores, a exposição contínua a reservatórios virais, como morcegos infectados, contribuiu para a difusão da doença. Mas, a partir das variações genéticas encontradas no vírus atual, concluiu-se que o surto de 2014 começou em uma única troca entre humanos, espalhando-se depois de pessoa para pessoa.
Os pesquisadores acham que o vírus foi para Serra Leoa a partir de duas linhagens do vírus originárias da Guiné. A possível fonte dessas linhagens foram 12 pessoas que participaram do funeral de um curandeiro na fronteira da Guiné, em maio.
Para chegar a essas conclusões, os cientistas estudaram amostras do vírus coletadas de 78 pacientes que foram diagnosticados com a doença em Serra Leoa nos primeiros 24 dias do surto. Alguns pacientes contribuíram com mais de uma amostra – o que permitiu estudar a mudança do vírus em cada indivíduo no decurso da infecção.
Para caracterizar as cepas atuais, os cientistas utilizaram a tecnologia conhecida como “sequenciamento profundo”, quando o procedimento é repetido inúmeras vezes para obter resultados de alta confiabilidade. No estudo da Science, cada genoma foi sequenciado em média 2 mil vezes, conforme os autores do artigo.
 RD – Jornal Repórter Diário Notícias sobre o ABC. Santo André, São Bernardo, São Caetano, Diadema, Mauá, Ribeirão Pires e Rio Grande da Serra
RD – Jornal Repórter Diário Notícias sobre o ABC. Santo André, São Bernardo, São Caetano, Diadema, Mauá, Ribeirão Pires e Rio Grande da Serra